
Bien que le sanglier (Sus scrofa) soit l’ancêtre du porc domestique (Sus scrofa domesticus), il existe des différences génétiques entre ces deux sous-espèces. Photo : Archives/TCN
Ce contenu est réservé aux abonné(e)s.
Pour un accès immédiat,
abonnez-vous pour moins de 1 $ par semaine.
S'abonner maintenant
Vous êtes déjà abonné(e) ? Connectez-vous
Le croisement entre le sanglier et le porc domestique produit un animal performant en termes d’élevage, mais qui perd les caractéristiques distinctives de la viande de sanglier. Pour la différenciation et le développement de marchés de niche, il est important d’authentifier les sangliers purs. Il est maintenant possible de le faire avec deux techniques en génétique, comme l’ont démontré les équipes des professeurs Allan King, de l’Université de Guelph, et Claude Robert, de l’Université Laval.
Bien que le sanglier (Sus scrofa) soit l’ancêtre du porc domestique (Sus scrofa domesticus), il existe des différences génétiques entre ces deux sous-espèces. Ces différences peuvent être révélées par deux approches, soit l’analyse de l’arrangement des chromosomes (caryotypage) et l’analyse du code génétique (génotypage). Pour répondre au besoin de l’Association des producteurs de sangliers du Québec (APSQ), qui désire confirmer l’appartenance génétique des animaux, un projet de recherche issu de la collaboration entre l’APSQ, le Centre de développement du porc du Québec et les professeurs Claude Robert, de l’Université Laval, et Allan King, de l’Université de Guelph, a été mis en œuvre pour évaluer l’efficacité de ces deux méthodes.
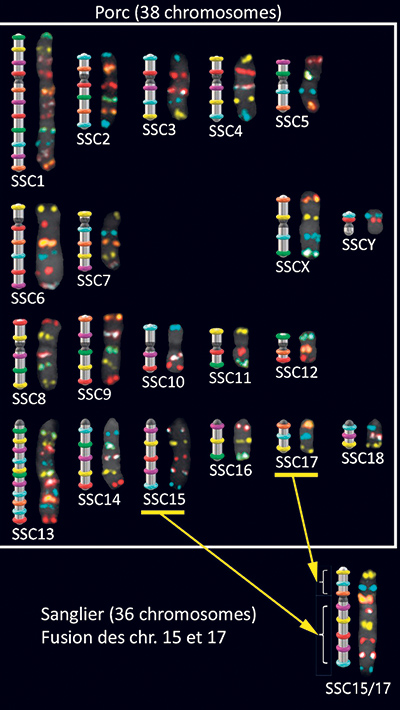
SSC1 à SSC18 : chromosome 1 à chromosome 18
SSCX : chromosome sexuel femelle X
SSCY : chromosome sexuel mâle Y
Adaptée de Poisson et coll., 2023.
Arrangement des chromosomes (caryotypage)
Le caryotypage est une méthode qui permet de visualiser l’arrangement des chromosomes et de compter ces derniers à partir d’une vue microscopique. Généralement, le nombre de chromosomes est le même pour tous les membres de la même espèce et le croisement interespèces n’est habituellement pas possible à cause d’un manque de complémentarité entre les deux jeux de chromosomes. Le sanglier est une des rares espèces qui échappe à la première règle. Le nombre théorique de chromosomes pour le sanglier d’Europe de l’Ouest a été défini à 36 et celui pour le sanglier d’Europe de l’Est et de l’Asie à 38. Or, en pratique, une grande variabilité est présente. Par exemple, l’analyse de 4 894 sangliers issus de populations sauvages et d’élevages en France a permis d’observer que 15,3 % d’entre eux possèdent 37 ou 38 chromosomes (Mary et coll., 2022). Jusqu’à maintenant, aucune analyse chromosomique de sangliers d’élevage au Québec n’avait été effectuée.
Le projet a permis d’analyser les chromosomes de 39 sangliers reproducteurs d’un troupeau de l’APSQ. Les caryotypes ont été réalisés au laboratoire de Karyotekk grâce à la collaboration du professeur Allan King. L’analyse chromosomique a révélé que 87,2 % des sangliers (34 sangliers sur 39) avaient 36 chromosomes, tandis que 10,2 % (4 sangliers) et 2,6 % (1 sanglier) avaient respectivement 37 (caryotype attendu pour un hybride) et 38 chromosomes (caryotype attendu d’un porc ou d’un sanglier asiatique). Ces résultats montrent qu’une variabilité du nombre de chromosomes est présente dans les populations de sangliers purs du Québec, au même titre qu’en Europe. Ce nombre variable de chromosomes est causé par la séparation d’un des chromosomes du sanglier en deux chromosomes comme c’est le cas chez le porc, ce qui ne modifie pas la quantité d’ADN de l’individu et n’affecte donc en rien la viabilité de l’animal ni ses caractéristiques physiques.
Puisqu’un porc normal a 38 chromosomes, l’analyse permet de confirmer qu’un animal porteur de 36 chromosomes n’est pas un porc. Par contre, il serait plus difficile de statuer sur l’appartenance des animaux ayant 37 ou 38 chromosomes, puisque des sangliers purs peuvent avoir ces nombres de chromosomes. La visualisation des chromosomes permet aussi la détection d’anomalies chromosomiques qui peuvent diminuer la fertilité des reproducteurs et se transmettre à la descendance. Chez le porc, après plusieurs années d’analyses chromosomiques, la fréquence de ces anomalies a été rapportée à 0,91 % de la population canadienne (King et coll., 2019). En ce qui a trait aux 39 sangliers soumis à l’analyse chromosomique, aucune anomalie n’a été détectée.
Code génétique (génotypage)
Les deux sous-espèces expriment des caractéristiques physiques différentes, ce qui implique que leurs codes génétiques diffèrent. Ce ne sont généralement pas des différences dans le nombre de gènes, mais plutôt des différences dans la séquence des gènes qui permettent une expression génétique différente. Même entre deux individus de la même espèce, il existe un grand nombre de variations fines de la séquence d’ADN. On retrouve souvent des variations d’une seule lettre un peu partout dans le génome, appelées SNP (Single Nucleotide Polymorphism ou « polymorphisme nucléotidique »). Le génotypage se base sur une connaissance préalable de plusieurs dizaines de milliers de ces SNP dans l’ADN. Le génotypage interroge alors chaque échantillon pour identifier les lettres de l’ADN pour ces milliers de SNP. Cela donne une description très fine de chaque génome permettant de distinguer des individus de la même famille. Les plateformes de génotypage sont appelées « puces à ADN ».
Pour le projet, la puce à ADN développée pour le porc, qui permet de déterminer les résultats d’environ 60 000 SNP (Illumina PorcineSNP 60), a été utilisée pour génotyper les mêmes sangliers que ceux soumis à l’analyse par caryotypage. Les données obtenues ont ensuite été comparées à celles de sangliers européens ainsi qu’à celles de porcs de races pures Duroc, Yorkshire et Landrace. Ces analyses ont été réalisées par l’équipe du professeur Claude Robert à l’Université Laval. Les données de génotypage ont permis de différencier les sangliers québécois des sangliers européens et des trois principales races de porcs utilisées au Québec. De plus, aucune distinction n’a été observée pour le regroupement génétique des sangliers à 36, 37 et 38 chromosomes, ce qui laisse croire que ces individus sont tous bel et bien des sangliers. L’utilisation d’un modèle basé sur l’intelligence artificielle a également permis de classer les sangliers et les porcs dans leurs groupes génétiques respectifs, et ce, sans intervention humaine.
En plus de permettre l’authentification des sangliers, les données issues du génotypage permettent d’obtenir plusieurs autres informations à partir d’analyses complémentaires. Par exemple, il est possible d’estimer la diversité génétique et la taille des effectifs génétiques de la population, soit le nombre d’individus suffisamment distincts génétiquement pour apporter une contribution au bassin génétique. Ce genre d’analyse requiert toutefois d’échantillonner une plus grande population afin d’avoir un comparatif et une idée du portrait global de la filière, ce qui n’a pas été possible dans le cadre de ce projet.
Toutefois, à la lumière des résultats obtenus, les producteurs de sangliers pourront développer un système d’authentification des animaux et même des produits de sanglier au besoin.
Remerciements
Ce projet est financé par l’entremise du Programme de développement sectoriel, en vertu du Partenariat canadien pour l’agriculture, entente conclue entre les gouvernements du Canada et du Québec, le Centre de développement du porc du Québec (CDPQ) et l’Association des producteurs de sangliers du Québec (APSQ). Ce projet a été réalisé grâce à la contribution des professeurs Allan King, de l’Université de Guelph, et Claude Robert, de l’Université Laval. Les auteurs remercient également les producteurs participants pour leur collaboration.
L’article a été rédigé en collaboration avec Richard Mailhot, conseiller technique, Centre de développement du porc du Québec, Allan King, professeur retraité, Université de Guelph, et Claude Robert, professeur, Université Laval.